Rows: 1000 Columns: 14
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (2): Sex, STRESS_EXIST
dbl (12): Age, Height, Weight, BMI, DM, HTN, Smoking, MACCE, Death, MACCE_da...
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.김진섭 대표는 1월 28일(월) 성균관의대 사회의학교실를 방문, tidyverse 생태계에서의 데이터 매니지먼트 방법을 강의할 예정입니다. 강의 내용을 미리 정리하였습니다.
시작하기 전에
R 데이터 매니지먼트 방법은 크게 3 종류가 있다.
data.table 패키지는 빠른 실행속도를 장점으로 tidyverse 의 득세 속에서 살아남았으며, 역시 과거 홈페이지2에 정리한 바 있다.
본 강의는 이중 두 번째의 기초에 해당하며
readr 패키지의
read_csv함수로 데이터를 빠르게 읽은 후magrittr 패키지의
%>%연산자와 dplyr 패키지의select,mutate,filter,group_by,summarize함수로 직관적인 코드를 작성하고purrr 패키지의
map함수로 쉽게 반복문을 처리하는 것을 목표로 한다.
각각의 패키지를 따로 설치할 수도 있고 install.packages("tidyverse")로 tidyverse 생태계의 패키지를 모두 설치할 수도 있다.
데이터 읽기: readr
readr 패키지에서 csv 파일을 읽는 함수는 read_csv이며, 구분자(ex: 공백, 탭)가 다를 때는 read_delim 함수를 이용하여 구분자를 설정할 수 있다. 예제 데이터를 읽어보자.
데이터를 읽으면 각 변수들을 어떤 형태(숫자형, 문자형)로 읽었는지 표현되는데, 바꾸고 싶은 것이 있으면 아래와 같이 col_types 옵션을 이용하면 된다.
## Character: col_character() or "c"
a <- read_csv("https://raw.githubusercontent.com/jinseob2kim/R-skku-biohrs/master/data/smc_example1.csv",col_types = cols(HTN = "c"))이제 데이터를 살펴보면 HTN 변수가 문자형이 된것을 볼 수 있다.
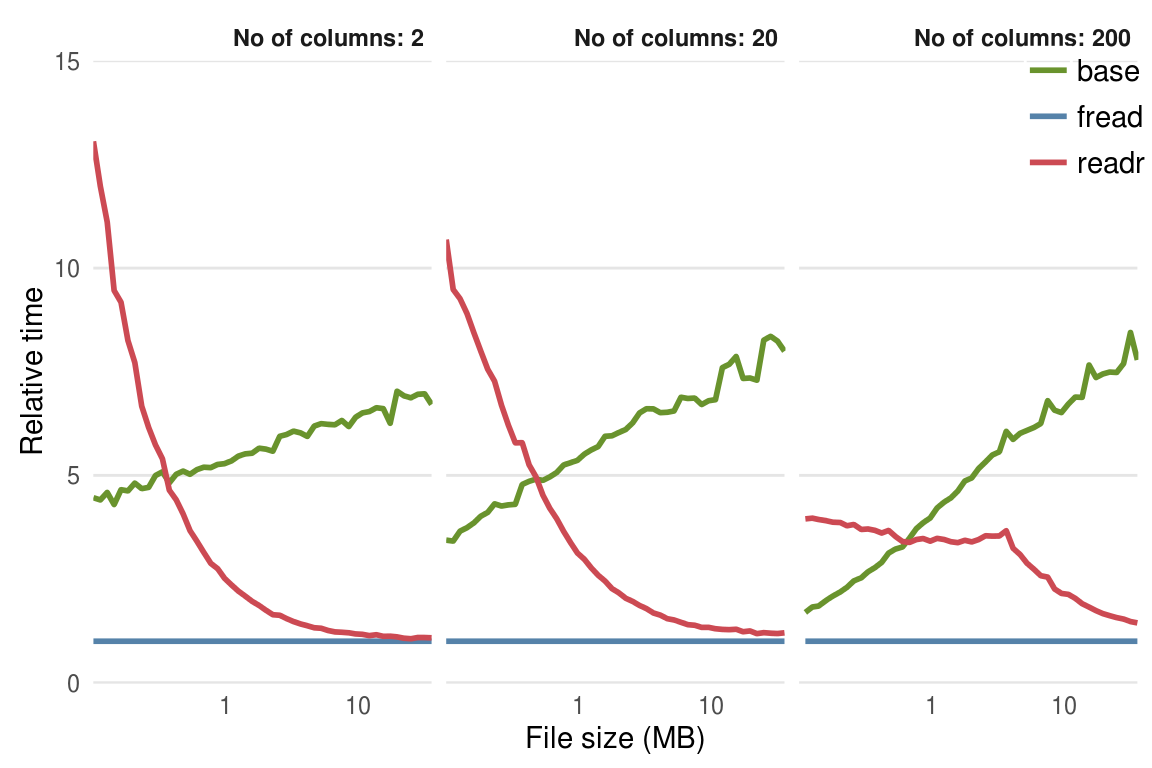
a기본 함수인 read.csv 와 비교했을 때 아주 작은 데이터에서는 장점이 없으나, 그 크기가 커질수록 read_csv가 더 좋은 성능을 보임이 알려져 있다(Figure @ref(fig:readfunc)).
read_csv로 읽은 데이터는 tibble이라는 새로운 클래스로 저장된다. 직접 확인해보자.
class(a)[1] "spec_tbl_df" "tbl_df" "tbl" "data.frame" 기존의 data.frame 외에도 tbl_df, tbl 와 같은 것들이 추가되어 있다. tibble은 data.frame보다 좀 더 날 것(?)의 정보를 보여주는데, 범주형 변수를 factor가 아닌 character 그대로 저장하고 변수명을 그대로 유지하는 것(data.frame에서 변수명의 특수문자나 공백은 .으로 바뀜)이 가장 큰 특징이다. tibble의 추가 내용은 jumpingrivers 블로그4를 참고하기 바란다.
직관적인 코드: %>% 연산자
tidyverse 생태계에서 가장 중요한 것을 하나만 고르라면 magrittr 패키지의 %>% 연산자를 선택하겠다. %>%은 Rstudio에서 단축키 Ctrl + Shift + M (OS X: Cmd + Shift + M)로 입력할 수 있는데, 이것을 이용하면 의식의 흐름대로 코딩을 할 수 있어 직관적인 코드를 작성할 수 있다. head함수를 통해 %>%의 장점을 알아보자.
head(a)와 a %>% head는 같은 명령어 “a의 head를 보여줘”로, a %>% head가 생각의 흐름이 반영된 코드임을 알 수 있다. 일반적으로 %>% 연산자는 함수의 맨 처음 인자를 앞으로 빼오는 역할을 하고 f(x, y)는 x %>% f(y)로 바꿀 수 있다. 맨 처음 인자가 아닐 때에는 y %>% f(x, .)과 같이 앞으로 빼온 변수의 자리에 .를 넣으면 된다. 예를 들면 아래에 나오는 세 명령어는 모두 같다.
%>%의 진가는 여러 함수를 한 번에 사용할 때 나타나는데, head와 subset을 동시에 쓰는 경우를 예로 살펴보겠다.
이 명령어는 a에서 남자만 뽑아서 head를 보여줘로 기존의 head(subset(a, Sex == "M"))보다 훨씬 직관적이며 함수가 3개, 4개로 늘어날수록 비교가 안될 정도의 가독성을 보여준다. 남자만 뽑아 회귀분석을 수행하고 그 계수와 p-value를 구하는 예를 살펴보자. 먼저 기존의 코딩 스타일대로 명령을 수행하면 아래와 같다.
다음은 %>% 를 이용한 코딩이다.
%>% 연산자로 쓴 코드는 읽기 쉬운 것은 물론 불필요한 중간변수(b, model, summ.model)가 없어 깔끔하고 메모리의 낭비도 없다. 딱 하나 주의할 점은 코드를 내려쓸 때 각 줄은 반드시 %>%로 끝나야 한다는 점이다. 예를 들어 아래의 코드를 실행하면 맨 윗줄만 실행되는 것을 확인할 수 있다.
a %>% subset(Sex == "M")
%>% head 본 강의 후 다른 것은 까먹어도 %>% 만 익숙해지면 성공이라고 생각한다.
데이터 정리: dplyr
dplyr 는 데이터를 효과적으로 다룰 수 있는 일련의 함수들을 제공한다. 이중 group_by와 summarize는 쉽게 그룹 별 요약통계량을 보여줌으로서 기존 R 문법과 차별화된 가치를 제공하므로 꼭 익혀두자.
filter
filter는 subset 함수와 같은 기능으로 특정 조건으로 데이터를 필터링하는 데 이용된다. 아래는 데이터에서 남자만 추출하는 예시이다.
filter에서는 & 외에 ,으로도 AND 조건을 쓸 수 있어 가독성이 좋다. between 함수를 이용하면 연속변수의 특정 범위를 선택할 수도 있는데, 이것 역시 기존의 &를 활용하는 것보다 직관적이다. 50~60세 사이를 필터링하는 예시를 살펴보자.
아래의 &나 ,로 표현한 조건도 between을 이용한 것과 같은 결과를 보여준다.
arrange: 정렬
arrange는 특정 순서에 따라 데이터를 정렬하는 함수로, 정렬 순서만 알려주는 order 함수와는 달리 정렬된 데이터를 보여주는 것이 특징이다.
## a[order(a$Age), ]
a %>% arrange(Age)정렬 조건이 2개 이상이면 ,로 같이 적을 수 있으며 내림차순 정렬은 desc 명령어를 이용한다. 아래는 Age에 대해 오름차순, BMI에 대해 내림차순 정렬을 수행하는 예시이다.
## a[order(a$Age, -a$BMI), ]
a %>% arrange(Age, desc(BMI))조건에 Age 대신 "Age"와 같이 문자열을 넣을 때는 언더바(_)가 붙은 arrange_ 함수를 이용하며, 이것은 나머지 함수들에서도 마찬가지이다.
a %>% arrange_("Age")
select: 변수 선택
select는 데이터에서 특정 변수들을 선택하는 함수로 기본 R 에는 없는 유용한 기능들을 제공한다.
## a[, c("Sex", "Age")]
a %>% select(Sex, Age, Height)변수명 대신에 열 번호를 넣어도 되며 Sex:Height을 이용해서 Sex와 Height 사이의 모든 변수를 선택할 수도 있다. arrange 함수와는 달리 "Sex"와 같은 문자열도 그대로 입력 가능하며, 아래의 방법들은 모두 a %>% select(Sex, Age, Height)와 같은 코드이다.
특정 변수들을 빼려면 -Sex나 -(Sex:Height)와 같이 적으면 된다.
## a[, -c("Sex", "Age", "Height")]
a %>% select(-Sex, -Age, -Height)아래의 코드도 a %>% select(-Sex, -Age, -Height)와 같은 결과를 준다.
만약 MACCE_date, Death_date와 같이 _date로 끝나는 변수들을 선택하고 싶다면 end_with 함수를 이용하면 된다.
## a[, grep("_date", names(a))]
a %>% select(ends_with("date"))이외에 select와 함께 쓸 수 있는 유용한 함수들을 정리하면 아래와 같다.
start_with("abc"): ’abc’로 시작하는 이름end_with("xyz"): ’xyz’로 끝나는 이름contains("ijk"): ’ijk’를 포함하는 이름one_of(c("a", "b", "c")): 변수명이a,b,c중 하나matches("(.)\\1"): 정규표현식 조건num_range("x", 1:3):x1,x2,x3
mutate: 변수 생성
mutate는 새로운 변수를 만드는 함수이다. Age와 BMI 변수에서 고령과 비만을 뜻하는 Old, Overweight 변수를 만들어 보자.
## a$old <- as.integer(a$Age >= 65); a$overweight <- as.integer(a$BMI >= 27)
a %>% mutate(Old = as.integer(Age >= 65),
Overweight = as.integer(BMI >= 27)
)새로운 변수만 보여주려면 mutate 대신 transmute를 사용한다.
a %>% transmute(Old = as.integer(Age >= 65),
Overweight = as.integer(BMI >= 27)
)
group_by와 summarize
group_by, summarize를 이용하여 원하는대로 그룹을 나누고 각 그룹의 요약통계량을 간단히 구할 수 있다. 기본 R에서는 aggregate함수가 같은 기능을 수행한다.
group_by에 "Age" 같은 문자열을 넣으려면 언더바(_)가 붙은 group_by_ 함수를 이용해야 한다.
모든 변수에 같은 요약방법을 적용하려면 summarize_all 함수를 사용한다. 아래는 50세 이상을 대상으로 모든 변수에 그룹별 평균을 적용한 예시이다.
평균값을 계산할 수 없는 범주형 변수는 NA를, 그 외 변수들은 평균값을 보여줌을 확인할 수 있다. 여러 요약값을 동시에 보여주려면 funs 명령어로 여러 함수를 같이 적어주면 된다.
%>%와 기본함수만으로 똑같이 구현하기.
마지막에 수행했던 작업을 %>%와 R 기본함수만으로 구현하면서 본 단원을 마무리하겠다. filter 대신 subset, select 대신 [], group_by와 summarize_all 대신에 aggregate를 이용하면 된다.
위와 같이 기본 함수로도 %>% 연산자를 이용하여 그럴듯하게 코드를 작성할 수 있으나, dplyr 와 비교했을 때 아무래도 가독성이 떨어진다는 점에서 dplyr가 유용한 패키지임을 확인할 수 있었다.
반복문 처리: purrr
본 내용에서는 for문이나 멀티코어의 사용은 언급하지 않는다. 해당 내용은 과거 강의5를 참고하기 바란다.
R에서 반복문을 처리하는데 가장 많이 이용되는 함수는 lapply(또는 sapply)일 것이다. purrr의 대표 함수인 map은 이 lapply를 tidyverse 철학으로 구현한 것이다. 이제부터 차이점을 알아보자.
map
데이터의 모든 변수들의 형태를 살펴보는 lapply 구문은 아래와 같다.
lapply(a, class)$Sex
[1] "character"
$Age
[1] "numeric"
$Height
[1] "numeric"
$Weight
[1] "numeric"
$BMI
[1] "numeric"
$DM
[1] "numeric"
$HTN
[1] "character"
$Smoking
[1] "numeric"
$MACCE
[1] "numeric"
$Death
[1] "numeric"
$MACCE_date
[1] "numeric"
$Death_date
[1] "numeric"
$STRESS_EXIST
[1] "character"
$Number_stent
[1] "numeric"이것을 map으로 구현하면 lapply와 정확히 같은 형태가 된다.
$Sex
[1] "character"
$Age
[1] "numeric"
$Height
[1] "numeric"
$Weight
[1] "numeric"
$BMI
[1] "numeric"
$DM
[1] "numeric"
$HTN
[1] "character"
$Smoking
[1] "numeric"
$MACCE
[1] "numeric"
$Death
[1] "numeric"
$MACCE_date
[1] "numeric"
$Death_date
[1] "numeric"
$STRESS_EXIST
[1] "character"
$Number_stent
[1] "numeric"map은 list 형태로 결과를 반환하며 특정 형태를 지정하려면 아래와 같은 함수들을 이용한다.
map: 리스트map_lgl: 논리값(T,F)map_int: 정수map_dbl: 실수map_chr: 문자열map_dfr:rbind된 data.framemap_dfc:cbind된 data.frame
앞의 예를 map_chr을 이용해 다시 실행하자.
map_chr(a, class) Sex Age Height Weight BMI DM
"character" "numeric" "numeric" "numeric" "numeric" "numeric"
HTN Smoking MACCE Death MACCE_date Death_date
"character" "numeric" "numeric" "numeric" "numeric" "numeric"
STRESS_EXIST Number_stent
"character" "numeric" 이것은 sapply함수의 tidyverse 버전으로 기억하면 좋다. 아래의 명령어들은 모두 같은 결과를 나타낸다.
각 변수에서 첫 번째 값을 뽑는 아래의 코드들도 sapply와 map_*의 차이는 없다.
Sex Age Height Weight BMI DM
"M" "52" "160" "63" "24.609375" "0"
HTN Smoking MACCE Death MACCE_date Death_date
"1" "1" "0" "0" "1056" "1056"
STRESS_EXIST Number_stent
"No test" "3" Sex Age Height Weight BMI DM
"M" "52" "160" "63" "24.609375" "0"
HTN Smoking MACCE Death MACCE_date Death_date
"1" "1" "0" "0" "1056" "1056"
STRESS_EXIST Number_stent
"No test" "3" a %>% map_chr(`[`, 1) Sex Age Height Weight BMI
"M" "52.000000" "160.000000" "63.000000" "24.609375"
DM HTN Smoking MACCE Death
"0.000000" "1" "1.000000" "0.000000" "0.000000"
MACCE_date Death_date STRESS_EXIST Number_stent
"1056.000000" "1056.000000" "No test" "3.000000" class나 mean, 그리고 `[` 등 간단한 함수들은 lapply를 쓰나 map을 쓰나 차이가 없다. map의 진가는 반복할 함수가 복잡할 때 드러난다. 성별로 같은 회귀분석을 반복하는 코드를 예로 들어 보자. 먼저 lapply를 이용한 코드는 아래와 같다.
[[1]]
Call:
lm(formula = Death ~ Age, data = x, family = binomial)
Coefficients:
(Intercept) Age
0.0461858 0.0001931
[[2]]
Call:
lm(formula = Death ~ Age, data = x, family = binomial)
Coefficients:
(Intercept) Age
-0.208833 0.004355 위 예시에서 알 수 있듯이 lapply에서 복잡한 함수를 반복하려면 function(x) 문이 꼭 필요하고 가독성을 저해하는 원인이 된다. 그러나 map에서는 ~로 간단히 function(x)를 대체할 수 있다. 아래 코드를 살펴보자.
[[1]]
Call:
lm(formula = Death ~ Age, data = ., family = binomial)
Coefficients:
(Intercept) Age
0.0461858 0.0001931
[[2]]
Call:
lm(formula = Death ~ Age, data = ., family = binomial)
Coefficients:
(Intercept) Age
-0.208833 0.004355 map함수를 이용하여 function(x)를 ~로, 성별 데이터에 해당하는 x를 .으로 바꾸니 훨씬 읽기가 쉬워졌다.
이번엔 같은 회귀분석을 수행한 후 Age의 p-value만 뽑는다고 하자.
[1] 9.016998e-01 2.094342e-07%>% 연산자를 이용해 나름대로 읽기 쉬운 코드가 되었다. 이것을 map_dbl로 다시 표현하면 아래와 같다.
[1] 9.016998e-01 2.094342e-07function(x) 가 없어 더 잘 보이기는 하나 고작 이 정도의 장점이라면 map을 쓸 필요가 없을 것 같다. map을 좀 더 적극적으로 사용해 보자.
[1] 9.016998e-01 2.094342e-07기존 코드는 .$coefficient와 .[8] 같은 부분이 거슬렸는데, map("coefficients"), map_dbl(8)으로 바꾸니 보기에 훨씬 깔끔하다. 참고로 map("coefficients") 은 map(`[[`, "coefficients")의, map_dbl(8)은 map_dbl(`[`, 8)의 축약형 표현이다. 사실 아까 다루었던 첫 번째 값 뽑기의 경우도 아래와 같이 더 간단하게 쓸 수 있다.
map을 통해 함수의 모든 중간과정을 따로따로 구현한 것도 장점이다. 에러가 발생했을 때 드래그로 중간과정까지만 실행할 수 있어 함수의 어느 부분에서 에러가 발생했는지를 쉽게 알아낼 수 있다.
map2, pmap: 입력 변수 2개 이상
2개 이상의 입력값에 대해 반복문을 수행하는 함수로는 mapply가 있다. 이것의 tidyverse 버전이 map2와 pmap인데 전자는 2개의 조건에, 후자는 리스트 형태로 입력값의 갯수에 상관없이 반복문을 구현할 수 있다. 본 강의에서는 간단한 예만 다루어 보겠다. 먼저 mapply를 이용해서 여러 입력값의 합을 구하는 코드를 살펴보자.
mapply는 첫번째 인수에 함수를, 그 다음부터는 입력값들을 2개, 3개… 계속 받을 수 있다. 반면 map2와 pmap은 입력값을 먼저 받는데, 이 때문에 pmap에서는 입력값들을 리스트 형태로 받는다.
map2(1:5, 1:5, sum)[[1]]
[1] 2
[[2]]
[1] 4
[[3]]
[1] 6
[[4]]
[1] 8
[[5]]
[1] 10pmap(list(1:5, 1:5, 1:5), sum)[[1]]
[1] 3
[[2]]
[1] 6
[[3]]
[1] 9
[[4]]
[1] 12
[[5]]
[1] 15리턴 형태를 지정하려면 map과 마찬가지로 map2_*나 pmap_* 꼴의 함수를 이용하면 되며 아래의 코드들은 같은 결과를 나타낸다.
마지막으로 paste함수로 문자열을 합치는 예를 살펴보겠다. 먼저 map2_chr로 두 문자열을 합쳐보자.
[1] "Alice was born at LA" "Bob was born at New york"첫 번째 입력값은 함수에서 .x로 두 번째 입력값은 .y로 표현할 수 있다. pmap 함수를 이용할 때는 ..1, ..2, ..3으로 바꿔 표현하면 된다.
마치며
지금까지 tidyverse 생태계에서 몇 가지 패키지를 이용해 데이터를 다루는 방법을 살펴보았다. 앞서 말했듯이 이 생태계에서 가장 중요한 것은 %>% 연산자를 이용하여 의식의 흐름대로 코딩을 수행하는 것이며, 이후 나머지 내용을 하나씩 적용해나가면 어느 순간 tidyverse 없이 살 수 없는(?) 자신을 발견하게 될 것이다. 본 글에서 다루지 않은 내용인 long, short 포맷을 다루는 tidyr, 문자열을 다루는 stringr, factor를 다루는 forcats, 날짜를 다루는 lubridate 그리고 모델을 다루는 modelr과 broom 등은 R for Data Science[^8]와 Rstudio cheetsheet[^9]를 참고하기 바란다. 다음번에는 빠른 속도가 장점인 data.table를 다시 한번 정리해 볼 생각이다.
Footnotes
Citation
@online{kim2019,
author = {Kim, Jinseob},
title = {R {데이터} {매니지먼트:} Tidyverse},
date = {2019-01-23},
url = {https://blog.zarathu.com/posts/2019-01-03-rdatamanagement/},
langid = {en}
}